Des plus hauts sommets aux fosses les plus profondes, on retrouve aujourd’hui des plastiques un peu partout dans notre environnement. Il s’agit évidemment d’un problème écologique de premier plan, dans la mesure où ils mettent traditionnellement un temps fou à se décomposer. La recherche travaille donc d’arrache-pied pour découvrir un moyen d’accélérer le processus; dans des travaux repérés par ScienceAlert, une équipe de l’Université du Texas vient de faire un grand pas dans cette direction en développant une enzyme capable d’y parvenir en quelques heures à peine.
Très sommairement, une enzyme, c’est une substance qui joue le rôle de catalyseur biologique; cela signifie qu’elle peut déclencher ou accélérer une réaction chimique. Ce sont des acteurs de premier plan du vivant; elles sont indispensables aux cycles de vie de la majorité des organismes vivants. On peut par exemple citer les protéases qui brisent les liaisons qui unissent les protéines, ou les lipases qui catalysent la décomposition des graisses.
Le PET décomposé en moins de 24 heures
Avec l’essor actuel de la bio-ingénierie, les chercheurs développent régulièrement des tas d’enzymes artificielles qui permettent de catalyser de nombreuses réactions chimiques indispensables à des processus industriels; on les trouve dans l’agroalimentaire, dans l’agriculture, l’industrie pharmaceutique…
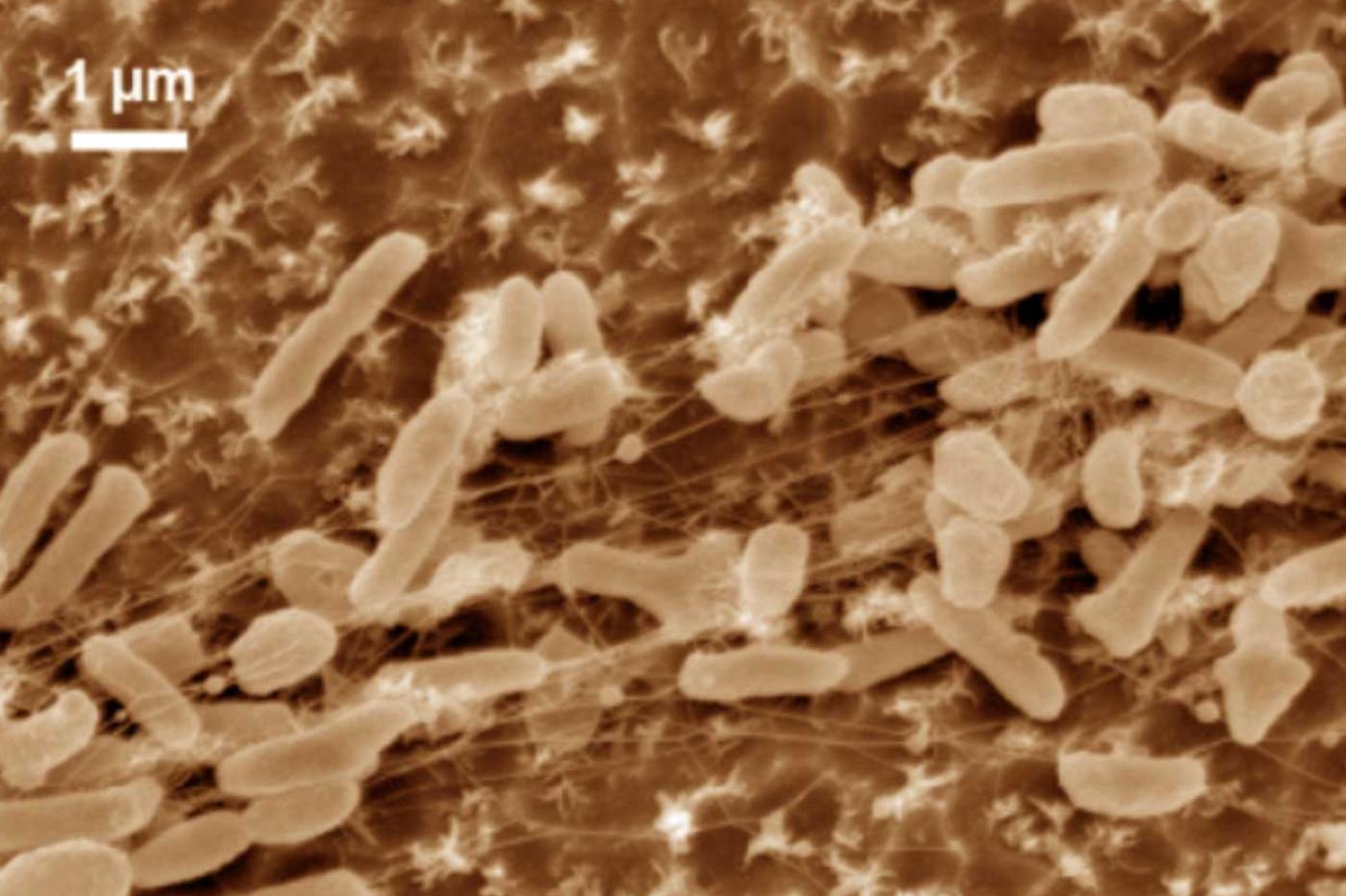
Les chercheurs sont ainsi partis d’une enzyme naturelle baptisée PETase. Comme son nom l’indique, elle est connue pour sa capacité à décomposer le plastique PET. Il s’agit d’un plastique parmi les plus utilisés, notamment dans les emballages qui finissent souvent dans la nature. Ils ont ensuite utilisé un système basé sur l’intelligence artificielle. Il leur a permis d’ identifier cinq mutations susceptibles d’augmenter l’activité catalytique de la PETase.
Cela leur a permis d’aboutir à une nouvelle enzyme, cette fois baptisée FAST-PETase pour Functional, Active, Stable and Tolerant PETase. Elle s’est révélée capable de catalyser la décomposition de ces plastiques en un temps record; selon les différents conteneurs testés, l’enzyme peut terminer sa tâche en quelques jours, voire même quelques heures à peine.
L’arsenal chimique d’une bactérie mangeuse de plastique
C’est un chiffre très impressionnant; rappelons qu’il faut habituellement des années, voire des siècles pour que ces plastiques se décomposent dans la nature. Et cette approche basée sur la FAST-PETase s’avère même plus rapide que toutes les autres techniques testées en ce moment.
De nombreux travaux ont par exemple été menés sur Ideonella sakaiensis. Il s’agit d’une bactérie découverte en 2016 qui a la particularité de pouvoir décomposer le PET pour s’en nourrir. Malheureusement, cette-ci met des dizaines de semaines à décomposer de petits échantillons moins résistants qu’une vraie bouteille en PET. Elle n’est donc pas adaptée à une utilisation à l’échelle industrielle.
En l’étudiant de plus près, les chercheurs ont cependant fini par découvrir l’élément responsable de cette faculté rare en 2016 : il s’agit justement de la fameuse PETase citée ci-dessus. Une fois isolée et purifiée, cette enzyme a permis d’accélérer considérablement le processus en décomposant le PET en quelques jours.
Enfin un recyclage à très grande échelle ?
Un chiffre qui commençait à devenir prometteur; et en le faisant passer sous la barre des 24 heures avec leur nouvelle FAST-PETase, l’Université du Texas vient potentiellement d’ouvrir la voie aux premières filières industrielles de recyclage enzymatique du plastique à grande échelle.
Le terme de recyclage n’est d’ailleurs pas choisi au hasard. Car la FAST-PETase ne se contente pas de réduire le plastique à l’état de bouillie inutilisable; elle le dépolymérise, c’est-à-dire qu’elle le découpe en petites unités qui peuvent ensuite être réassemblées. Les chercheurs ont d’ailleurs prouvé qu’il était possible de produire de nouveaux plastiques à partir des produits de la dégradation !
De plus, cette enzyme peut fonctionner à température ambiante, ce qui constitue un avantage très important en conditions réelles. On se retrouve donc avec une approche particulièrement prometteuse pour l’industrie, mais aussi et surtout pour l’environnement. En effet, le fait de recycler davantage plastique signifie qu’il y en aura moins qui sera entassé ad vitam æternam dans des décharges ou brûlé, avec tout ce que cela implique en termes de dépenses d’énergie et de toxicité.
“Ces travaux démontrent la puissance d’une approche multidisciplinaire, avec des notions tirées de la biologie synthétique, de l’ingénierie chimique et de l’intelligence artificielle”, se félicite le biochimiste Andrew Ellington.
🟣 Pour ne manquer aucune news sur le Journal du Geek, abonnez-vous sur Google Actualités. Et si vous nous adorez, on a une newsletter tous les matins.









Génial. Je suis impatient de ne plus jamais en entendre parler, comme a chaque fois qu on nous sort LA solution a un problème
c’est pas faux Snorgl… comme on compte meme plus les batteries révolutionnaires jamais sorti
Pendant ce temps en France ont va développer l’usine de retraitement pour 2025 https://www.franceculture.fr/economie/carbios-le-recyclage-enzymatique-du-plastique
Et le dit procédé de décomposition entraine-t-il des émissions polluantes ou à effet de serre-livres?
Rappelons qu’au depart le PET a ete invente, developpe et financé parce que entièrement recyclable sur lui meme ! Ou est passé cette affirmation qui a abusé le monde des plastiques ? Et pourqoui fait on encore des recherches pour tourner en rond ?
Si l’enzyme part dans un cours d’eau, elle va ronger tous les plastiques du monde… Ballot…